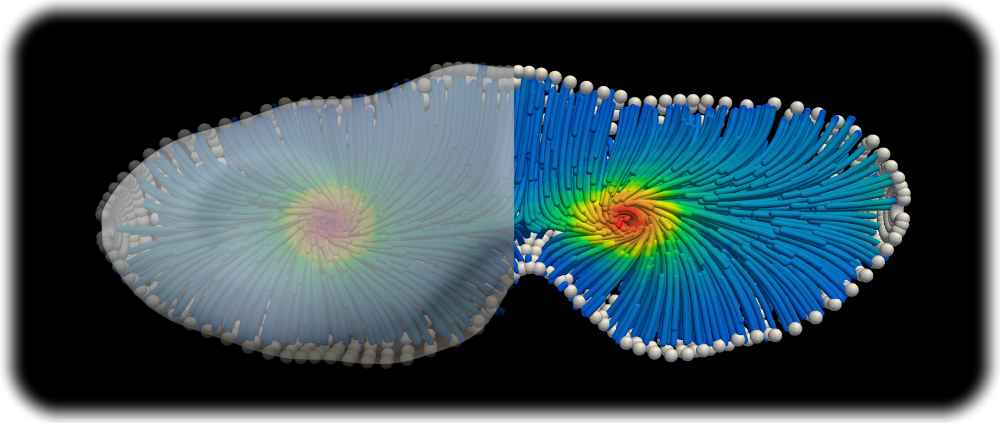
3D Simulation eines aktiven Materials in einer geometrischen Form, die einer sich teilenden Zelle ähnelt. Abb.: Singh u.a. in: Physics of Fluids (2023) via MPI-CBG
Rechner können nun simulieren, wie sich Zellen und Organe in Raum und Zeit formen
Dresden, 21. November 2023. Dresdner Forscher haben einen Code geschrieben, mit dem sich die Entwicklung „aktiver Materie“ in Lebewesen auf Supercomputern simulieren lässt. Damit haben sie einen wichtigen Schlüssel geliefert, wie Zellen, Organe und andere biologische Strukturen entstehen. Das geht aus einer Mitteilung des Max-Planck-Instituts für molekulare Zellbiologie und Genetik (MPI-CBG) in Dresden hervor.
Schlüssel zu künstlichen Bio-Maschinen
„Unser Code eröffnet neue Möglichkeiten, aktive Materialien zu erforschen“, betonte Prof. Ivo Sbalzarini, einer der Studienautoren. „Damit könnten wir endlich verstehen, wie Zellen und Gewebe ihre Form erhalten und damit die grundlegende Frage der Morphogenese beantworten, die Wissenschaftlerinnen und Wissenschaftler seit Jahrhunderten fasziniert. Der Code könnte uns auch dabei helfen, künstliche biologische Maschinen mit minimaler Anzahl von Komponenten zu entwerfen.“
Video zur Supercomputer- Simulation (MPI-CBG):
Zellmotoren wandeln Phosphor-Kraftstoff in Bewegung – und strukturieren dabei die Materie
Hintergrund: In menschlichen und tierischen Zellen sind winzige biologische „Motoren“ für den Transport von Materialien und Energieträgern zuständig. Diese Motoren wandeln Kraftstoffe wie das Molekül „Adenosintriphosphat“ (ATP) in Bewegung um. „Auf diese Weise entstehen Bewegungsmuster, und das Material strukturiert sich durch ständigen Energieverbrauch selbst“, erklären die CBG-Forscher. Derart selbst organisierende Materialien seien auch als „aktive Materie“ bekannt. Die Mechanik von Zellen und Geweben lasse sich insofern mit einer Theorie der aktiven Materie beschreiben.
Krankhafte Deformationen bald leichter voraussagbar
Die Vorgänge in aktiver Materie mathematisch zu erfassen, war indes seit jeher eine Herausforderung. Wissenschaftlerinnen und Wissenschaftler des CBG, des benachbarten „Zentrums für Systembiologie Dresden“ (CSBD) und der TU Dresden haben nun aber einen Algorithmus dafür entwickelt. Diese Beschreibung haben sie in einen quelloffenen Supercomputer-Code übersetzt, „der die Gleichungen der Theorie aktiver Materie zum ersten Mal in realistischen Szenarien lösen kann“, heißt es vom CBG. „Diese Ergebnisse bringen uns der Lösung des jahrhundertealten Rätsels, wie Zellen und Gewebe ihre Form erhalten und dem Design künstlicher biologischer Maschinen einen großen Schritt näher.“ Möglich sei es damit, das Verhalten aktiver Materie dreidimensional im Raum sowie in der Zeit zu simulieren. „Dank unserer Arbeit können Wissenschaftlerinnen und Wissenschaftler jetzt zum Beispiel die Form eines Gewebes vorhersagen oder wann ein biologisches Material instabil oder fehlreguliert wird“, erklärte Studienautor Philipp Suhrcke von der TU Dresden. „Das ist bedeutend, um die Mechanismen von Wachstum und Krankheit besser zu verstehen.“
An der Entwicklung dieser Theorie der aktiven Materie und der darauf basierenden Supercomputer-Programme waren unter anderen Abhinav Singh und Philipp H. Suhrcke von der TU Dresden, Prof. Ivo Sbalzarini vom CSBG, Direktor Prof. Frank Jülicher vom Max-Planck-Institut für Physik komplexer Systeme Dresden und CBG-Direktor Prof. Stephan Grill beteiligt.
Quelle: MPI-CBG
Wissenschaftliche Publikation:
„A numerical solver for active hydrodynamics in three dimensions and its application to active turbulence“ von Abhinav Singh, Philipp H. Suhrcke, Pietro Incardona und Ivo F. Sbalzarini, in: „Physics of Fluids“ 35, 105155 (2023), Fundstelle im Netz: DOI 10.1063/5.0169546
Der Computercode dieser Studie ist im Github-Verzeichnis 3Dactive-hydrodynamics verfügbar unter: https://github.com/mosaic-group/3Dactive-hydrodynamics
Die Open-Source-Bibiothek OpenFPM ist verfügbar unter: https://github.com/mosaic-group/openfpm_pdata

Ihre Unterstützung für Oiger.de!
Ohne hinreichende Finanzierung ist unabhängiger Journalismus nach professionellen Maßstäben nicht dauerhaft möglich. Bitte unterstützen Sie daher unsere Arbeit! Wenn Sie helfen wollen, Oiger.de aufrecht zu erhalten, senden Sie Ihren Beitrag mit dem Betreff „freiwilliges Honorar“ via Paypal an:
Vielen Dank!

Du muss angemeldet sein, um einen Kommentar zu veröffentlichen.